-
Compteur de contenus
1 605 -
Inscription
-
Dernière visite
Tout ce qui a été posté par Jean-Luc Bethmont (Picroformol)
-

Melosira auxospores...suite
Jean-Luc Bethmont (Picroformol) a posté un sujet dans Diatomées (Collections)
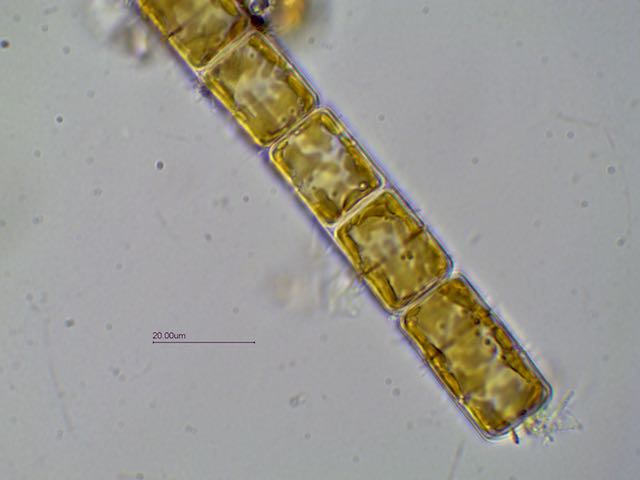
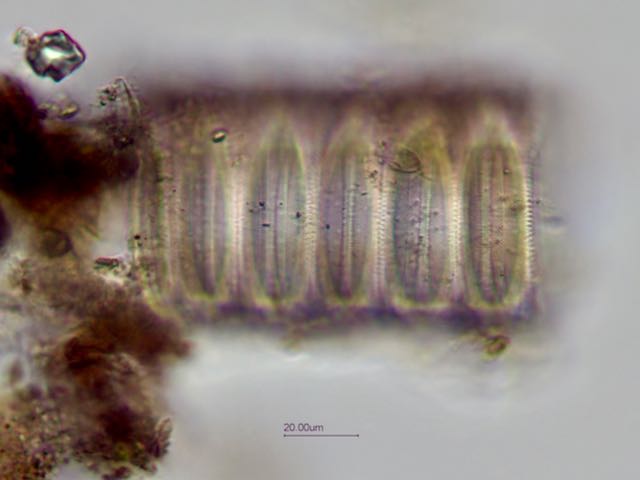
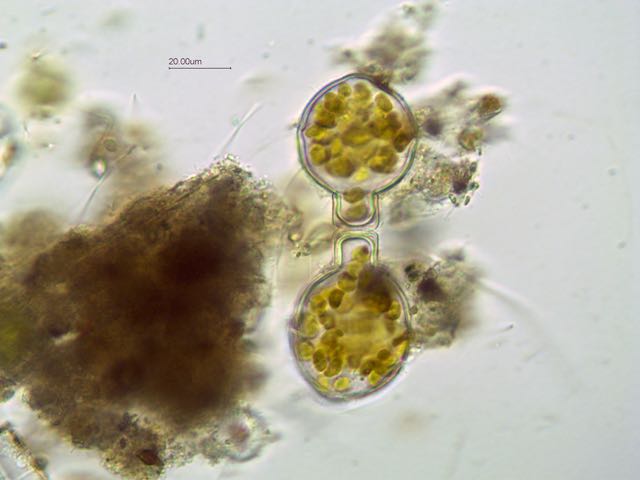
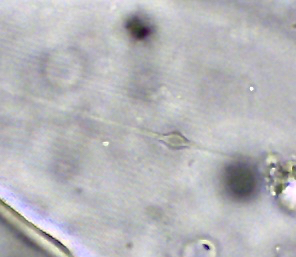
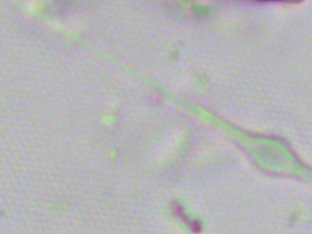
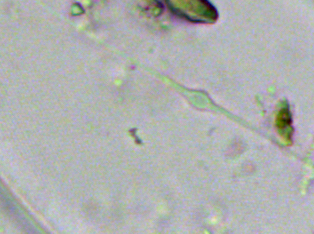
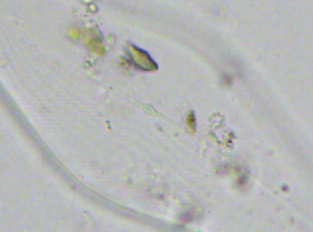
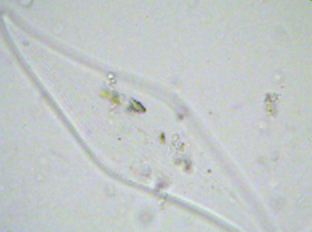
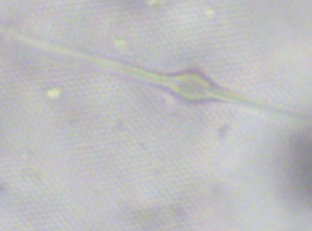
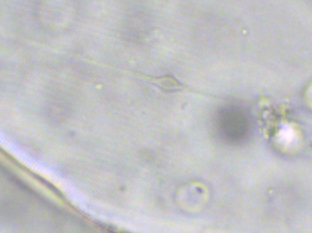
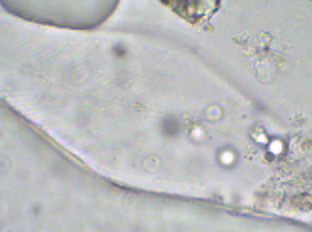
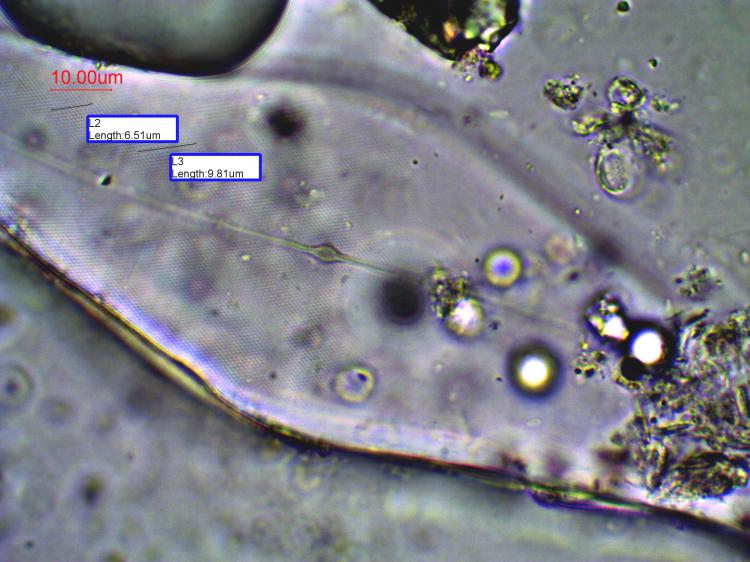
Je ne sais pas pourquoi mais on ne peut pas répondre à ce sujet et j' ai mis des tailles de photos trop petites donc je fais une suite avec la bonne photo: On voit mieux les détails. Le frustule mesure environ 60 µm de diamètre et 20 µm d 'épaisseur. Cordialement, JL -
Bonjour, Présente en assez grande quantité dans un échantillon d' eau douce (mare dans le Val d' Oise) la diatomée Melosira forme des filaments: Deux autres vues x60 et x100: Au niveau des frustules vides ont aperçoit des "dents" à droite permettant l' amarrage de chaque frustule et les stries qui ornementent chaque thèque: enfin les auxospores qui correspondent à des oeufs fécondés (reproduction sexuée) Sur le site suivant une expication des modes de reproduction: https://sites.google.com/site/sheobiogt/organism-study/life-cycle Cordialement, JL
-

Foie Foie gras Stéatose
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Merci pour ces précisions -

Foie Foie gras Stéatose
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Encore une belle présentation. J' ignorais qu' il pouvait y avoir des hépatocytes avec des noyaux polyploïdes; est-ce qu'il y en a dans les noyaux de cellule d' autres organes et celà a-t-il un rapport avec la cancérogénèse ? Amicalement, JL -

Plume oiseau biologie
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonsoir Dominique, Encore un sujet enrichissant. Je suis toujours stupéfait par les capacités d' adaptation du vivant via les mutations . Amicalement, JL -

Logiciel photo et camera
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Erhelge dans QUESTIONS POSEES PAR LES VISITEURS A MIKOSCOPIA
Bonjour Eric, Question délicate ! Il y a quelques années j' ai acheté une caméra Optixcam Summit serie 5mp qui était donnée compatible mac le modèle équivalent est dispo ici: https://www.microscope.com/microscope-cameras/optixcam-summit-sk2-5-2x-5-0mp-pc-mac-compatible-digital-microscope-camera.html En fait la caméra est compatible mac mais le logiciel de traitement de l' image ( mesure, échelle, annotation) ne l' est pas. j' ai donc acheté d' occasion un pc sous windows XP SP3 que je laisse à côté du microscope. Donc il faut faire attention aux compatibilités indiquées D' autre part le fait d' utiliser un port USB2 entre la caméra et l' ordinateur limite, pour la vidéo, le nombre d' image par seconde; je plafonne à environ 15 image/sec à une résolution de 1280x960 ce qui est insuffisant pour les organismes rapides. Par exemple la caméra ci dessous est compatible mac et pc y compris pour le logiciel et en usb 3 je ne connais pas le prix c' est un modèle plutot pro. https://www.microscope.com/microscope-cameras/advanced-professional/gryphax-subra-2-0-mp-hd-cmos-color-digital-microscope-camera.html Sinon il y a des caméras autonomes qui n' ont pas besoin d' ordi (stand alone) qui enregistrent sur carte sd et la visu se fait sur écran via cable hdmi comme le modèle ci dessous: https://www.microscope.com/omano-ocs-hdmi-1080pu-2-0mp-hdmi-usb2-0-digital-microscope-camera.html Enfin je fais tourner quelques logiciels spécifiques ( iris,picolay) avec " wine" sur le mac. Cordialement, JL -

Philippe Labrot Ingénieur
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Tryphon T dans MON COIN LABO
Merci pour l' info. Une présentation très intéressante des techniques microscopiques et les photos qui vont bien avec ! Cordialement, JL -

Lyrella lyra
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Discussions)
Il y a peut-être une solution avec des logiciels de stacking qui utilisent des "cartes de profondeur" et, de là, font une représentation 3D de l' objet (voir Picolay par exemple) cordialement, JL -

Microtome à main
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de jynxtrop dans Préparations microscopiques des échantillons
Bonjour Jl, Regarde l' article suivant: http://www.microscopies.com/DOSSIERS/Magazine/Articles/WD-Tranchette/Tranchette.html Et fait quelques essais Cordialement, JL -

Coscinodiscus lewisianus
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Discussions)
Merci Pierre Effectivement le rendu est plus "mou" -

Coscinodiscus lewisianus
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Discussions)
Bonjour Pierre, Encore une petite merveille! Pourquoi ne mets-tu pas une photo en éclairage normal comme "référence" ? Cordialement, JL -

Surirella tenera
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Collections)
Bonjour Pierre, J' aime bien la dernière photo où le pont cytoplasmique est bien visible. Cordialement, JL -

Kittonia granulata 4 : vue cingulaire
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Collections)
Bonjour Pierre, Singulière vue cingulaire ! (désolé je n'ai pas pu m' en empêcher) On se demande comment elles peuvent se détacher ? Cordialement, JL -

Stauroneis gracilis
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Collections)
Merci pour la dédicace. J' ai en préparation un autre "petit monde" Cordialement, JL -

Etude des diatomées test : 1. Frustulia rhomboides
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Collections)
Hello Pierre, Très intéressant mais je suis étonné par la première photo pourquoi n' a t-on pas une meilleure résolution ? Comment as tu réglé le diaphragme d' ouverture (à quelle ouverture?) -

Microtome de ranvier sur AllSciences
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Clément Tondo dans Préparations microscopiques des échantillons
Bonjour Clément, Je te suggère de lire l' article suivant si tu veux t' entraîner à faire des coupes à peu de frais: http://www.mik-mag.fr/2004/05/tips-techniques-sur-lutilisation-de-la-tranchette-a-deux-lames-de-rasoir/ JL -

Module à diaphragme pour Aristoplan
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Marc Babalian dans Leitz LEICA
bonjour Jean Marc, Je suis aussi admiratif devant un tel projet mené à bien. cordialement, JL -

auliscus eximius
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de pablito dans Diatomées (Collections)
Bravo tu maîtrise ! Le Père Noël est passé par là JL -

Kittonia elaborata : une diatomée marine fossile peu commune
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de patrice duros dans DIATOMEES
Splendide diatomée! Sait-on à quoi servent ces protubérances ? Cordialement, JL -

Feuille Histologie Consistance
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Encore un sujet original et bien documenté, bravo! Il m' inspire la réflexion suivante: derrière la diversité macroscopique des feuilles il y a des schémas d' organisation assez restreints et des "matériaux de construction" communs. La nature aime bien "bricolée" autour de plans éprouvés. Cordialement, JL -

Recyclage d'un Spectroscope.
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Tryphon T dans Sujets de Makers
Bonjour Tryphon, Je vais essayer de répondre à tes interrogations (je ne sais pas exactement dans quel but cet appareil a été construit): oui . Si tu n' a qu'un seul capteur tu ne pourras faire qu' une seule mesure à une longueur d' onde donnée à la fois. Evidemment en déplaçant le capteur ou en tournant le réseau tu feras défiler les longueurs d' ondes et réaliser un spectre par exemple pour identifier une substance. Lorsque l'on parle de spectrométrie (mesure de spectre) on ne précise pas si il s' agit de spectre d'émission ou de spectre d' absorption. La spectrométrie d' émission: Par exemple l' atome Fer présent dans certaines étoiles (le soleil entre autre) est chauffé par le soleil (excité) les électrons en revenant sur leurs orbites émettent une lumière d' une longueur d' onde caractéristique. Suivant le niveau d' excitation il peut y avoir différentes raies d' émission; L' ensemble de ces raies forme le spectre d' émission du fer (atomique). on peut ainsi faire une recherche d' éléments dans une étoile (présence ou absence) La spectrométrie d' absorption: Dans ce cas on utilise une lampe pour éclairer la substance (ion , molécule) qui est en solution dans un solvant (eau ou autre) contenu dans une cuve en verre ayant un trajet optique bien défini (en général 1cm) et des faces parallèles. En faisant des mesure d' absorptions à différentes longueur d' onde (exprimées en D.O. =densité optique) on réalise un spectre d' absorption). Concernant le Fer (à ma connaissance) on ne peut pas le mettre en solution sous forme atomique c' est donc l' ion Fer (Fe2+ ou Fe3+) qui se retrouve en solution (je n' ai aucune idée du spectre?). En général pour caractériser un ion tel le Fer on le fait réagir avec un composant spécifique il forme alors un complexe coloré On peut faire de la spectrophotométrie en infrarouge, dans le domaine visible et en ultraviolet. On peut faire également de la spectrométrie d' absorption atomique.........Bon j' arrête je vais boire l' apéro Cordialement, JL -

Le coin labo de Vardar
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Vardar dans MON COIN LABO
Bonjour Phil, Je suis admiratif (et envieux) c' est plus qu' un coin...labo???? -

Test Capteur Resolution
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Luc Bethmont (Picroformol) dans Appareils de prise de vue
En fait je me suis rendu compte après coup que j' avais recadré les photos en imposant un nombre de pixels pour hauteur et largeur alors que, pour mieux comparer, j' aurais dû imposer une hauteur et largeur fixe en unité de longueur(P.E. (500 µm sur 400 µm). Jérôme voici la photo 2 traitée (contraste, luminosité, etc...) on voit bien les trous: Photo 3 c' est du moiré photo 7 j' hésite la voici ci dessous: Je pensais que le moiré arrivait si on suréchantillonnait de trop ? Dans cette démarche j' ai choisi le fait que le capteur ignore certains pixels pour obtenir les échantillonnages inférieurs il faudrait aussi faire la manip avec le binning car chaque "superpixel" créé recevrait plus de lumière ce qui diminuerait le bruit. Cordialement, JL -

Test Capteur Resolution
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Luc Bethmont (Picroformol) dans Appareils de prise de vue
Voici les photos prises dans les même conditions qu' au départ pour: L' objectif x60 recardées en 312x232 pixels fréquence capteur 227 (OK) L' objectif x60 recardées en 312x232 pixels fréquence capteur 114 (OK) L' objectif x60 recardées en 312x232 pixels fréquence capteur 57 (Insuffisant ) L' objectif x60 recardées en 312x232 pixels fréquence capteur 28 (Insuffisant) L' objectif x100 recardées en 312x232 pixels fréquence capteur 227 (OK) L' objectif x100 recardées en 312x232 pixels fréquence capteur 114 (OK) L' objectif x100 recardées en 312x232 pixels fréquence capteur 57 (Insuffisant mais on aperçoit qq stries) L' objectif x100 recardées en 312x232 pixels fréquence capteur 28 (Insuffisant) Pour bien se rendre compte du résultat il faut augmenter le contraste des photos et les remettre aux mêmes dimensions en cm; Bien qu' il soit perfectible ce test permet de se faire une idée de la réponse du capteur. cordialement, JL -

Test Capteur Resolution
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Luc Bethmont (Picroformol) dans Appareils de prise de vue
Bonjour, Dans mon message de départ je n' avais pas pu continuer mon test avec les objectifs x60 et x100 à immersion pour cause de problème de distance de travail trop courte avec la préparation. J' ai enfin trouvé un autre sujet: une diatomée récupérée sur des coquilles d' huitres mise entre lame et lamelle dans l'eau. Je pense que c' est pleurosigma angulatum. Le nombre moyen de stries pour 10 µm va de 18 à 22 (Maurice Loir); J' ai mesuré une distance moyenne entre 2 aérolations de 0,65 µm soit un peu plus de 15 stries pour 10 µm: Cette photo permet d' avoir une vue d' ensemble (obj. x100) le contraste a été rehaussé pour mieux visualiser les aérolations Les autres photo sont dans le message suivant