-
Compteur de contenus
1 605 -
Inscription
-
Dernière visite
Tout ce qui a été posté par Jean-Luc Bethmont (Picroformol)
-

Crabe - cœur - anatomie histologie
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Bravo pour cette superbe présentation qui a dû de demander pas mal de temps ! cordialement, JL -

Ananas structures histologiques
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Merci Dominique pour toutes ces informations sur l' ananas. Le vocabulaire des botanistes est vraiment riche, précis et varié ! Je l' apprécie aussi dans la Piña colada. Amicalement, JL -

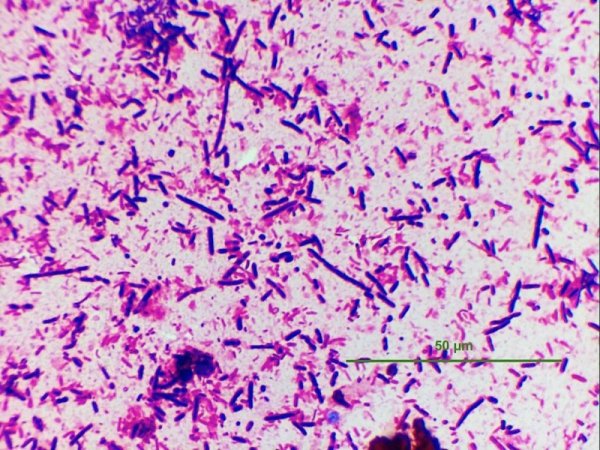
Bacillus Megaterium.jpeg
Jean-Luc Bethmont (Picroformol) a posté une image de la galerie dans Galerie Mikroscopia
-

Se faire un microscope pour 28€
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de jmp76 dans Sujets de Makers
Super bonne idée 👍 -

Capture Rapide
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Luc Bethmont (Picroformol) dans Appareils de prise de vue
Bonjour, Merci pour vos conseils, j'ai aussi retrouvé un document de Jean Marc " Modifier l’éclairage classique d’un microscope en éclairage LED". Je vais regarder aussi comment commander la LED avec les ports GPIO du Raspberry et comment integrer cela avec le "DC-DC step down de JMP" . Comme je n'y connais pas grand chose en électronique cela va me demander un certain temps.... Je reviendrais vers vous pour d' autres conseils. Amicalement, JL -

Capture Rapide
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Luc Bethmont (Picroformol) dans Appareils de prise de vue
Bonjour à tous, D' abord mes excuses Jean-Marie ! JMP j' ai relu ton article que j' avais oublié: il est toujours bon de s' inspirer de l' expérience des autres... Voici les photos pour la place de la lampe dans mon micro: cette première photo montre la lampe dans son boitier retirée du statif (lampe 6v 30W) la deuxième vue de profil il y a 12 mm de dégagement: la troisième l' arrière du statif ou vient s' enclencher le boitier avec les emplacements des deux grosses fiches qui maintiennent le boitier: Est ce qu'une LED de ce type peut convenir https://www.e44.com/composants/composants-actifs/optoelectronique/led/leds-cms/led-puissance-3w-star-blanc-ambiant-750ma-3300k-3.4...4v-LED3WCMS-WW.html et comment l' alimentée pour avoir un courant continu filtré avec une tension commandée via les ports GPIO du Raspberry ? P.S. Concernant ChatGPT: il me rend de grands services pour l'écriture de code avec ses limites bien sûr ! Pour Neat Image il m'a cité une bibliothèque Python qui ferait aussi bien que neat Image c'est "bm3d" je ne l'ai pas encore testée. Amicalement, JL -
Bonjour Dominique, J'ai lu ton sujet, j' avais l' impression de lire un roman policer. Tu mènes une véritable enquête... Amicalement, JL
-

Capture Rapide
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Luc Bethmont (Picroformol) dans Appareils de prise de vue
Bonsoir Jean-Marc, Je te remercie pour ton intérêt et je pense quand réunissant l' informatique, les micro ordi (Arduino,Raspberry Pi etc...) et nos microscopes ont pourrait faire pas mal de projets intéressants. Concernant le zooplancton il n'y a pas de problème, la détection doit aussi fonctionner il suffit d' ajuster le paramètre de seuil de la détection. Dans mon exemple j' utilise l' aire calculée par le contour de l' organisme en mouvement quelque soit sa vitesse et sa forme. La bibliothèque OpenCV est riche, on peut détecter des carrés ,des ronds, des structures particulières (coins) sans compter sur l' utilisation de modèles entraînés (deep learning) etc... Concernant le scintillement j' avais pensé aux LEDs mais je n'ai pas les connaissances nécessaires pour faire un tel montage (remplacer la lampe halogène du micro par une LED) et effectivement l' idée de suralimenter la LED est très intéressante. La RaspberryPi possède des broches GPIO permettant de piloter pas mal de choses. Le programme pourrait avoir la séquence suivante: détection, suralimentation LED, prise de vue, arrêt de la suralimentation de la LED. Si tu peux faire un schéma de montage avec alimentation de la LED, les caractéristiques des pièces à commander, je suis preneur... Enfin on peut gagner en luminosité en faisant un binning 2x2 ou 4x4 par addition (regroupement des valeurs des pixels) ce qui multiplie par 4 ou 16 la luminosité (au détriment du nombre de pixel constituant l' image. Image neat (que je n' utilise pas encore) améliore le bruit et préserve les détails. Je vais demander à chatGPT comment il travail afin d' essayer de l' ajouter au programme (c'est peut-être ambitieux !) Amicalement, JL -

Capture Rapide
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean-Luc Bethmont (Picroformol) dans Appareils de prise de vue
Suite...... Les photos sont prises à l' objectif x40 et recadrées. La vitesse de prise de vue va du 1/2000 au 1/1000. On arrive à obtenir des détails intéressants ! Il y a pas mal de déchets dans les photos, il faut faire du tri après. Comme le temps de pose est très court je suis obligé d' augmenter le gain ce qui produit du bruit. l' autre problème vient du courant alternatif qui alimente l' éclairage halogène (50 Hz) ce qui fait qu'il y a du "scintillement" (flickering) en desous d' un temps d' expo inférieur au 1/100. Je dois refléchir à ces 2 points...... Mais c'est quand même cool de lancer le programme et d' aller boire un café en attendant Cordialement, JL -
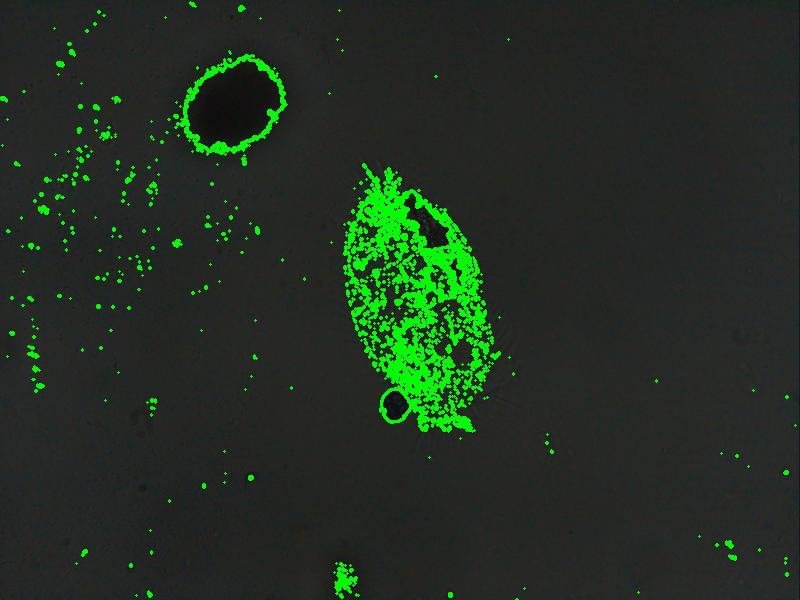
Bonjour, Pour rebondir sur le sujet du ralentissement des ciliés et des remarques de Jean-Marc qui utilise un flash incorporé à l' éclairage du microscope je me suis demandé si l' on pouvait tiré le portrait de ces "bolides" avec une caméra basique. J' utilise maintenant un capteur Sony IMX477 pilotable entièrement via la bibliothèque Picamera2 (en python) avec un Raspberry Pi4: https://forum.mikroscopia.com/topic/19143-camera-sony-et-raspberry/#comment-79351 Donc j' ai développé un programme qui permet de capturer des images avec une vitesse d' obturation de 100 à 1000 µ secondes (soit 1/10000 à 1/1000 sec). Pour pouvoir déclencher automatiquement la prise de vue j' ai choisi de détecter les objets (ciliés) en mouvement dans le champ de capture sur la base de la soustraction du fond (background subtractor). Je met sur la platine la lame avec l' échantillon et la lamelle, je fais la mise au point et je démarre le programme. Une première photo du fond est capturée puis le programme recherche à chaque capture combien de pixels ont changés. Le programme trace le contour des objets en mouvement , si l' objet en mouvement à une surface dont la valeur est supérieure à une limite alors la prise de vue est déclenchée et la photo est enregistrée puis le programme attend un nouvel objet en mouvement etc... Ci dessous l' écran de contrôle de détection du mouvement: Pour avoir plus de chance de détecter un cilié j' utilise un morceau de lamelle. Voici quelques ciliés détectés:
-

Talle chez certaines poacées Blé - Orge - Plateau de tallage
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Ton article est, encore une fois, très interessant (mes parents cultivaient du blé en autre) effectivement les rendements ont fortement augmentés depuis 1950. Je voulais juste ajouter que la récolte dépend de nombreux facteurs en dehors d'un talle de bonne qualité comme les conditions météo avant la récolte (blé couché qui ne peut être moissonné) épis et grains chétifs ,moisissure etc... Amicalement, JL -

Cochenille les dégâts sur les branches .
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour, J' ai trouvé ce site sur les cochenilles: https://passion-entomologie.fr/cochenilles-vecteurs-de-virus/ Cordialement, JL -

Pholcus phalangioïde
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Tu nous racontes là une belle histoire (naturelle). A quoi correspond la zone verdâtre au sommet de la tête ? Cordialement, JL -

Planctons d'automne 2024
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean Marie Cavanihac dans Jean Marie CAVANIHAC
Merci pour ta réponse mais je m' aperçois que la durée de conservation est assez réduite ! Donc profitons de tes images ... -

Planctons d'automne 2024
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Jean Marie Cavanihac dans Jean Marie CAVANIHAC
Bonjour Jean Marie, Quel beau bestiaire tu nous offres là ! J' ai une question: il m' arrive d' aller de temps à autre au bord de mer, je fais des prélèvements que je ramène à la maison pour observation mais ils ne se conserve pas. Quel est le moyen le plus simple pour améliorer cette conservation afin d' avoir le temps de poursuivre les observations et si il faut des produits spécifiques où se les procurer. Merci d' avance, JL -

Spectromètres
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Tryphon T dans Sujets de Makers
Bonjour Tryphon, Quel est le domaine d' utilisation des spectromètres de poche ? J' ai utilisé des spectrophotomètres de laboratoire dans le domaine de l' analyse médicale mais concrètement que fait-on avec un spectro de poche ? Cordialement, JL -

Coléoptère élytres ailes
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Une fois de plus on voit comment la "nature" est ingénieuse ! Comment as -tu réalisé cette coupe ? B.c. JL -

ortie et les filatures
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Encore un sujet bien documenté et qui ne manque pas de piquant :) cordialement, JL P.S. je me demande si l' ortie était cultivée avec les moyens agronomiques actuels sa culture resterait "écologique" -

Calamar le sytéme circulatoire
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Encore un beau sujet ! Est ce qu' il y a des éléments figurés qui circulent dans ce système ? Cordialement, JL -

Calamar encre anatomie histologie
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Encore un superbe travail. Concernant les produits retrouvés dans l' encre: Je pense que le plomb et le cadmium sont des polluants accumulés par l'organisme (je ne sais pas à Quelle concentration ils sont ?) Cordialement, JL -

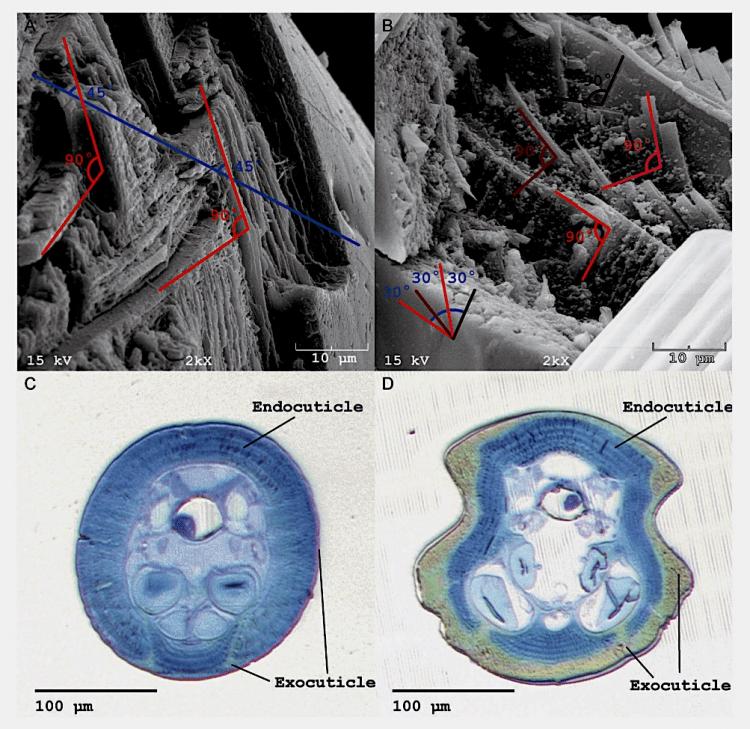
Charançon trompe rostre
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Concernant la dureté de la chitine du rostre du charançon j' ai trouvé cet article ou on donne une explication (il vaut mieux que son rostre soit solide car il n' en a qu' un seul pour toute sa vie :) ) : https://asknature.org/?s=weevil&page=0&is_v=1#video et une photo avec une coupe du rostre similaire a ton schéma: Cordialement, JL -

Crevettes 2 - éléments d’anatomie -
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour Dominique, Sujet encore bien documenté qui a dû te demander beaucoup de travail. Je suis toujours admiratif de la qualité de tes photos ! Cordialement, JL -

Thymus Ris de veau
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Bonjour, Effectivement je me souviens d' un documentaire sur le sujet où il était question d' études qui mettaient en évidence le fait que des personnes qui avaient côtoyé des animaux dans leur enfance étaient moins sujet aux allergies et étaient plus "robustes" vis à vis des infections. Cordialement, JL -

Coquille d’ escargot histologie
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Dominique. dans Dominique
Encore un article enrichissant. Et pas une coquille dans le texte........ Cordialement, JL -

Automate de coloration.
Jean-Luc Bethmont (Picroformol) a répondu à un(e) sujet de Tryphon T dans Sujets de Makers
Bonjour Tryphon, Superbe réalisation. Je pense que le système fait monter/descendre la lame dans les flacons et que les flacons sont sur un rotor ? Pour les coupes botaniques ont pourrait envisager un panier (métallique ou plastique) pour les transporter d' un colorant à l' autre . Cordialement, JL





.jpeg.b403325ad477bc2164623d34e5bfda43.jpeg)
.jpeg.e968a806660824874ccde969cf26f06e.jpeg)
.jpeg.a2b4494f51e9c31eb20314ce8b8c5da7.jpeg)
.jpeg.1f8a1a019b2e794e0c65990ac92056ae.jpeg)
.jpeg.4b469939fcc97ea0461d444b9b9459ab.jpeg)
.jpeg.53bc86c40ef083734ed0c12364937413.jpeg)
.jpeg.5de29e4bb1bbdebe5a5f5148b04e7ae3.jpeg)
.jpeg.887425898ee9656cf9f71b5d0c4f079f.jpeg)